Wie funktionieren Proteine, durch die ein Bakterium zum Krankheitserreger wird? Und wie injizieren Bakterien die krankheitserregenden Moleküle in menschliche Zellen? Mit diesen Fragen befassen sich der Biochemiker Professor Dr. Hartmut Niemann von der Universität Bielefeld und seine Arbeitsgruppe. Das in vielen krankheitserregenden Bakterien zu findende Sekretionssystem (T3SS) schießt Virulenzproteine durch eine winzige Nadel in Wirtszellen. In einer Studie untersuchten die Forschenden diesen molekularen Injektionsapparat von Yersinia enterocolitica, einem Erreger für Magen-Darm-Beschwerden. Die Studie wird heute (02.06.2022) in der Fachzeitschrift Nature Communications vorgestellt.
Der Forschungsschwerpunkt von Professor Dr. Hartmut Niemann ist die Strukturbiochemie. Sein Ziel ist es, die dreidimensionale Struktur von Proteinen zu bestimmen. Die Arbeitsgruppe von Niemann ist auf Virulenzproteine von krankheitserregenden Bakterien spezialisiert. Virulenzproteine sind spezielle Moleküle, durch die ein Bakterium zu einem Krankheitserreger wird. So gibt es zum Beispiel bei dem bekannten Darmbakterium E. coli Stämme, die harmlos für den menschlichen Körper sind, und weitere Stämme, die krankheitserregend sind.
„Bei einigen Bakterien haben sich ausgefeilte Strukturen entwickelt, mit denen sie gezielt Moleküle ausschleusen und in Zellen des Menschen injizieren können, um eine Infektion auszulösen“, sagt Hartmut Niemann. Diese Strukturen werden als Typ-III-Sekretionssysteme (T3SS) bezeichnet. Die molekularen Injektionsapparate sind in vielen krankheitserregenden Bakterien zu finden, wie zum Beispiel bei Yersinia enterocolitica, einem Erreger relativ harmloser Magen-Darm-Beschwerden. Das Bakterium ist vor allem bei Schweinen verbreitet und vom Tier auf den Menschen übertragbar.
Zu finden sind Typ-III-Sekretionssysteme auch bei den durchfallverursachenden Salmonellen und bei Pseudomonas aeruginosa, einem antibiotikaresistenten Krankenhauskeim, der chronische Infektionen der Atemwege verursacht. „Der Sekretionsapparat ist bei vielen bakteriellen Krankheitserregern ähnlich, aber nicht identisch“, so Niemann. „Unterschiede zwischen den einzelnen Bakterien bestehen vor allem darin, welche Proteine sie mit dem Sekretionssystem in die Zellen des Wirts transportieren.“
Herausfinden, wie krankmachende Proteine im Bakterium ausgewählt werden
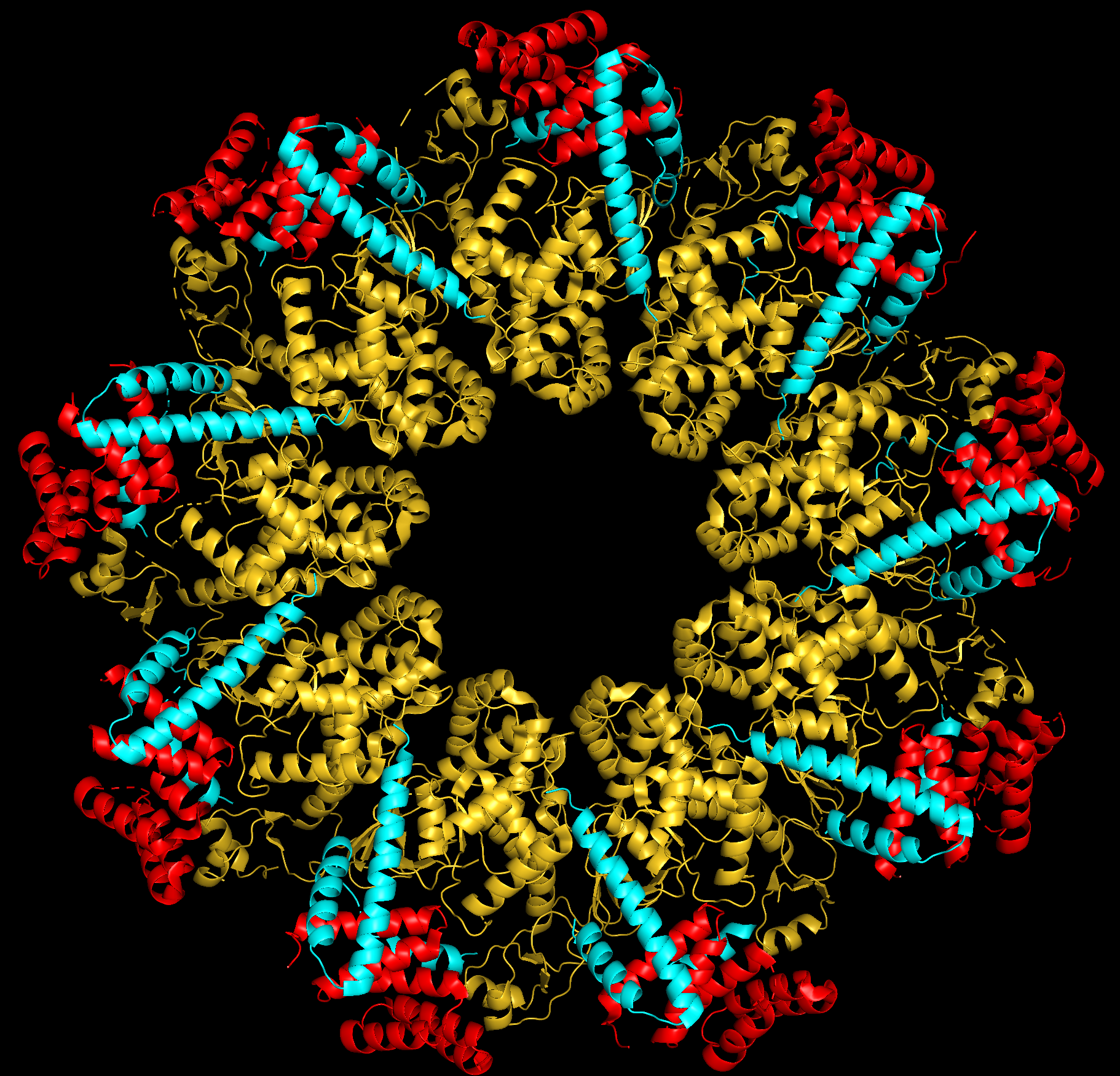
Die Sekretionssysteme bestehen aus verschiedenen Komponenten, die in einer festen Reihenfolge aus den Bakterien heraus transportiert werden müssen, damit eine gezielte Reaktion ausgelöst wird. Der Frage, wie T3SS kontrollieren, welches Protein sie während einer Infektion in welcher Reihenfolge transportieren, widmete sich Dominic Gilzer in seiner Promotion bei Hartmut Niemann. Gilzer ist wissenschaftlicher Mitarbeiter in Niemanns Arbeitsgruppe. Ziel seines Dissertationsprojekts war es, zu verstehen, wie ein Protein, das an der Einhaltung der richtigen Sekretionsreihenfolge beteiligt ist, an das Sekretionssystem bindet, bevor es selber ausgeschleust wird.
Gilzer hat gemeinsam mit seiner Kollegin Madeleine Schreiner und mit Hartmut Niemann einen Bestandteil des Sekretionssystems untersucht: den Export-Apparat. Dieser ist für die Auswahl der krankmachenden Proteine im Bakterium und für deren Einschleusen in die Injektionsnadel des Sekretionssystems verantwortlich. Der Apparat besteht aus mehreren Proteinen, von denen das Studienteam eines genauer erforschte: Es handelt sich um den Teil des Export-Apparats, der die Virulenzproteine erkennt, die in die Wirtszellen transportiert werden sollen.
Zusätzlich untersuchte die Arbeitsgruppe ein weiteres Protein. Es ist an den Export-Apparat gebunden und wird ebenfalls nach außen transportiert. „Dieses Protein ist interessant, weil es für die Funktion des Sekretionssystems von Yersinien absolut notwendig ist. Bisher ist aber unklar, zu welchem Zweck es ausgeschleust wird und ob es nach dem Verlassen des Bakteriums noch eine Funktion erfüllt“, sagt Dominic Gilzer. „Möglicherweise ist nur wichtig, dass es den Export-Apparat nicht länger blockiert und damit Bindeplätze für andere Proteine freigibt“, erklärt er. Gilzers Arbeit zeigt jetzt erstmals, wie ein Protein vor dem Ausschleusen mit dem ringförmigen Export-Apparat von Yersinien wechselwirkt. Hartmut Niemann zieht als Fazit: „Der Vergleich unserer Ergebnisse mit früheren Arbeiten deutet darauf hin, dass der Andockpunkt für Proteine, die ausgeschleust werden sollen, in den Export-Apparaten verschiedener Sekretionssysteme sehr ähnlich ist. Diese Vermutung gilt es noch weiter zu erforschen.“
Mit Röntgenkristallographie Moleküle des Proteins erkennen
Um die dreidimensionale Struktur des Proteins zu erfassen, nutzte das Studienteam die Röntgenkristallographie. Dies ist die älteste und am besten etablierte Methode, mit der fast alle Proteine untersucht werden können. Diese Methode ermöglicht es, die räumliche Struktur eines Proteins zu bestimmen, also die einzelnen Atome in dem Molekül und ihre Verbindungen untereinander sichtbar zu machen. In der Röntgenkristallographie werden die Proteine kristallisiert, um sie im zweiten Schritt mit Röntgenstrahlen zu beschießen.
Die Proteinkristalle dienen bei der Methode lediglich als Verstärker zur Ablenkung der Röntgenstrahlen. „Würden wir mit einem Röntgenstrahl ein einzelnes Molekül beschießen, würden wir nichts sehen. Deshalb nehmen wir einen Kristall, in dem Hunderte von Millionen oder Milliarden Moleküle regelmäßig angeordnet sind“, sagt Niemann. „Wir machen Grundlagenforschung, um zu verstehen, wie die T3-Sekretionssysteme funktionieren. Wir hoffen, dass unsere Ergebnisse anderen Forschungsgruppen helfen, Wege zur molekularen Hemmung dieser Systeme zu finden und so Infektionen zu unterdrücken“, erläutert Hartmut Niemann. „Unsere Motivation für diese Grundlagenforschung ist, dass die T3-Sekretionssysteme eine große Rolle für viele bakterielle Krankheitserreger spielen.“
Die Forschungsdetails der Arbeitsgruppe und ihre Ergebnisse sind in Nature Communications veröffentlicht worden. Nature Communications ist eine begutachtete wissenschaftliche Open-Access-Fachzeitschrift, die Arbeiten zu allen wissenschaftlichen Themengebieten publiziert – insbesondere aus den Naturwissenschaften, vor allem der Biologie, der Physik, der Chemie und den Geowissenschaften. Das Journal liegt mit seinem Impact-Faktor auf Rang 3 von 64 wissenschaftlichen Zeitschriften in der Kategorie „multidisziplinäre Wissenschaften“.