Bei manchen Infektionen zählt jede Minute, um ein Leben zu retten: Kommen Patient*innen mit einer Sepsis ins Klinikum, müssen Mediziner*innen schnellstmöglich entscheiden, welches Antibiotikum sie geben – oft ohne den genauen Erreger oder seine Antibiotikaresistenz zu kennen. Die Wahl fällt dann meist auf ein breit wirkendes Medikament. Das birgt Risiken. Hier setzt das seit Oktober 2022 durch das Bundesministerium für Gesundheit geförderte und nun abgeschlossene Projekt KINBIOTICS an.
Für das erste wissenschaftliche Projekt, in dem alle im neuen Universitätsklinikum vereinten Kliniken zusammen mit der Uni Bielefeld zusammengearbeitet haben, zieht Professor Philipp Cimiano, Initiator und Leiter des Projektes, eine positive Bilanz. Ein erstes Ziel war die Entwicklung eines KI-Systems, das anhand von Parametern, die über die Patient*innen bekannt sind, errechnen kann, welches Antibiotikum das wahrscheinlich wirksamste sein wird. Die Grundlagen dafür lieferte unter anderem die von Anästhesist und Intensivmediziner Professor Sebastian Rehberg geleitete Universitätsklinik für Anästhesiologie, Intensiv-, Notfallmedizin, Transfusionsmedizin und Schmerztherapie am Evangelischem Klinikum Bethel (EVKB).
„Bei der Auswahl der Antibiotika müssen wir immer mehrere Faktoren berücksichtigen, zum Beispiel den Erreger, den Infektionsort und die Begleiterkrankung des oder der Patient*in“, erklärt der Mediziner. Grundlagen für diese Entscheidung sind alle bekannten Patient*innendaten: Laborwerte, Notfallbefunde, Vorerkrankungen, Erkenntnisse zu neuen Resistenzen oder zu Wirkmechanismen der Antibiotika. „Das KI-System kann uns da helfen, in dem es automatisiert diese ganzen Daten abgleicht und auswertet, um dann Therapievorschläge zu machen. Die Entscheidung trifft aber letztlich immer noch ein Mensch.“
Um das System zu trainieren, stellen die teilnehmenden Kliniken zum einen die notwendigen medizinischen Daten zur Verfügung. Zum anderen werden die Ergebnisse, die das System in der Praxis lieferte, überprüft. „Wir sind auf einem guten Weg“, sagt Sebastian Rehberg. „Wir sind aber noch nicht so weit, dass es uns im Alltag maßgeblich hilft.“

© Universität Bielefeld
Mit dem bislang Erreichten ist auch Projektleiter Philipp Cimiano sehr zufrieden: „Wir haben es mit Daten aus den drei Kliniken des Klinikums OWL geschafft, ein KI basiertes Modell zu trainieren und erfolgreich zu evaluieren.“ Das Modell sei außerdem in ein Nutzungssystem eingebettet worden, das von Ärzt*innen in den drei Häusern getestet wurde. „Die Rückmeldung war sehr positiv. Damit ein solches System aber zugelassen würde, müsste eine klinische Studie durchgeführt werden.“ Für Sebastian Rehberg ist es aber auch ein Erfolg, dass die drei Kliniken so gut zusammengearbeitet haben. „Es ist eines der ersten größeren Projekte dieser Art. Dadurch haben sich fachübergreifende Strukturen entwickelt, die jetzt auch für andere Projekte genutzt werden.“
Das zweite Ziel des Projektes war die Beschleunigung der Identifizierung von Erregern: „Das ist eines der großen Probleme in der klinischen Praxis“, sagt Philipp Cimiano. Mit der für das Projekt entwickelten schnelleren Erregerdiagnostik kann diese Zeit von mehreren Tagen auf rund zwei Stunden reduziert werden. „Diese schnellen Erregertests werden im Evangelischen Klinikum Bethel (EVKB) schon experimentell genutzt.“

© Universität Bielefeld
Entwickelt hat dieses System das Team um Professor Jörn Kalinowski, am CeBiTec unter anderem Leiter der Technologieplattform Genomik. „Wenn innerhalb von Minuten entschieden werden muss, wäre es doch fantastisch zu wissen, welche Antibiotika man nicht gibt“, sagt er. „Die Erregeridentifikation läuft über eine DNA-Analyse, die in unserem Fall sehr schnell sequenziert und auch identifiziert“, erklärt Jörn Kalinowski. Möglich machen das kleine Sequenzier-Geräte, die mit einem etwa Laptop-großen Computer verbunden sind. „Die Software vergleicht die Sequenzen mit den gespeicherten Sequenzen bekannter Erreger und zeigt dann anhand der genetischen Struktur, wogegen die getesteten Erreger resistent sein könnten.“ Getestet wurde die neue Technologie im Institut für Laboratoriumsmedizin, Mikrobiologie und Hygiene am EVKB. „Das ist noch in den Kinderschuhen, es lassen sich so aber Infektionsketten schneller entdecken – innerhalb von Stunden, weil nicht erst Blutkulturen angesetzt werden müssen“, sagt die Leiterin des Instituts, Oberärztin Dr. Christiane Scherer.
Nun muss die Technik noch schneller werden, um die Daten auch im Notfall rechtzeitig bereitzustellen. „Schon jetzt zeigt sich aber das Potenzial, das die Technik hat“, erklärt Jörn Kalinowski. Zukünftig soll auch die Funktion, Antibiotika vorzuschlagen, implementiert werden. Für Jörn Kalinowski steht fest: „Wir machen auf jeden Fall weiter.“

© Universität Bielefeld
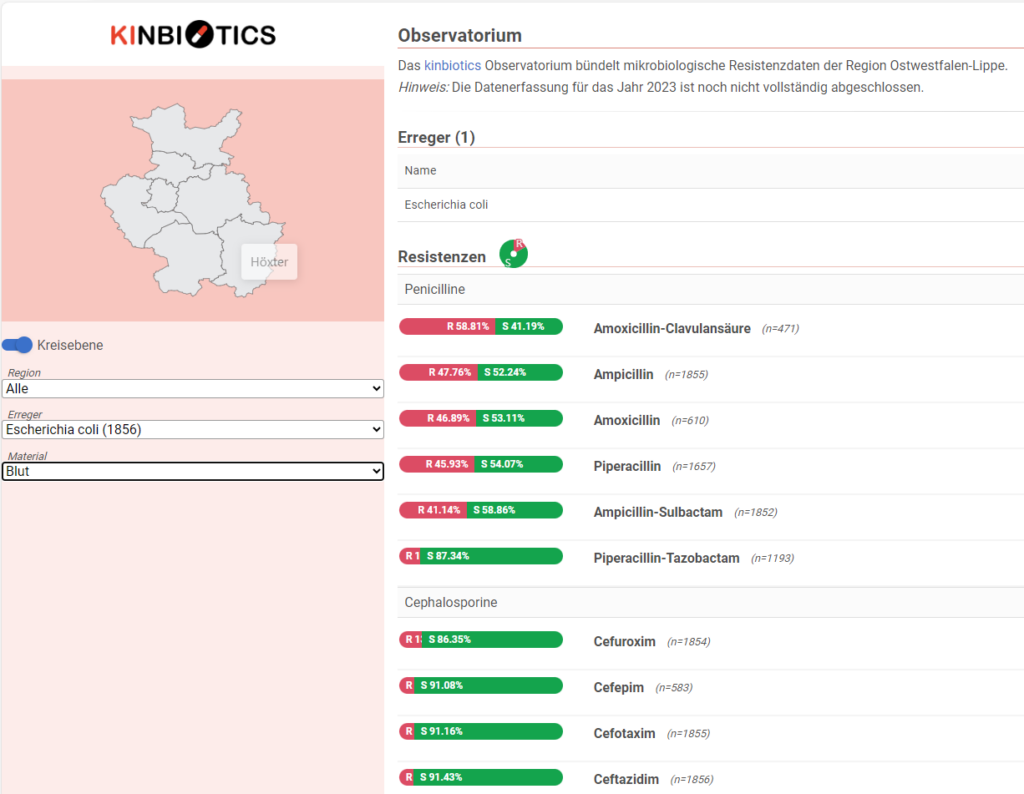
Als drittes Ziel sollte ein OWL-weites Resistenz-Observatorium entwickelt werden, um das lokale Auftreten von Erregern und Resistenzen zeitnah abbilden zu können. „Wir haben ein Observatorium entwickelt, in dem die Daten aus den verschiedenen Krankenhäusern in einen integrierten Datenbestand zusammengefügt werden“, erklärt Projektleiter Philipp Cimiano. „Hier können auch niedergelassene Ärzt*innen sehen, wie die Erregerlage gerade ist – das gab es so vorher nicht. Damit zeigen wir auch, dass ein intensiver Datenaustausch über die Häuser hinweg gewinnbringend ist.“ Er und sein Team hoffen, dass hier künftig auch die Daten von weiteren Laboren einfließen, die zum Beispiel im Auftrag der Hausarztpraxen erstellt werden.
Auch am Aufbau des Resistenz-Observatoriums ist das Institut für Laboratoriumsmedizin, Mikrobiologie und Hygiene am EVKB beteiligt. „Wir liefern mit den Kliniken Bielefeld Mitte und Lippe die Daten zu den Erregerresistenzen, um die KI zu trainieren“, erklärt Christiane Scherer. Diese Daten der drei beteiligten Häuser wurden bislang weder strukturiert zusammengefasst, noch zeitnah veröffentlicht. „Die Daten des EvKB werden zwar laufend über eine Schnittstelle an das RKI übermittelt, dort werden sie aber nur jährlich aktualisiert und nur grob regional aufgeschlüsselt.“ Viel zu selten, um so das Infektionsgeschehen oder die Entwicklung von Resistenzen zu beobachten. „Mit dem Resistenz-Observatorium können wir das nun viel zeitnaher, lokaler und auch materialspezifischer betrachten.“

© Universität Bielefeld
Da derzeit nicht alle Labore in OWL Teil des Projektes sind, sind die Daten bislang nur bedingt aussagekräftig. „Die Idee ist, dass nicht nur im Krankenhaus tätige, sondern auch niedergelassene Ärzt*innen schauen können, wie die aktuelle Resistenzlage aussieht, um zu entscheiden, ob sie ein Standardantibiotikum oder ein anderes verordnen“, sagt Christiane Scherer. „Die Entwicklung resistenter Bakterienstämme könnte man so früher erkennen und sich darauf vorbereiten.“
© Universität Bielefeld
Für Projektleiter Philipp Cimiano bedeutet das Projektende nicht das Ende der Forschung: „Im Moment beantragen wir ein Folgeprojekt beim Bundesministerium für Gesundheit, ein kürzeres Projekt, in dem wir auf Basis der Erkenntnisse von KINBIOTICS eine App entwickeln wollen, die allen Mediziner*innen in Deutschland zur Verfügung steht.“ Darüber hinaus ist geplant, eine möglichst nationale Datenbank aufzusetzen, um bessere KI-Modelle zu entwickeln. „Wir werden an allen Stellen weitermachen. Alle Ergebnisse sind praxisrelevant, es ist aber nicht so, dass man sie morgen schon in die Praxis integrieren kann – dafür sind noch weitere Schritte notwendig.“